查看更多

 明星产品
明星产品
 干货推荐
干货推荐
 关注我们
关注我们

扫码关注我们
了解更多信息
上一篇干货分享中我们全面介绍了Cas系统,小伙伴们已经对CRISPR/Cas9系统的基础原理进行了初步掌握,而基因敲除作为CRISPR/Cas9技术最为广泛的应用,相信大家对于具体怎样实现基因敲除会感到十分好奇,小编这就来带大家系统的了解一下CRISPR/Cas9系统在基因敲除方向的应用,本篇干货主要介绍内容包括基因敲除原理、敲除单克隆细胞株的构建、基因片段敲除和敲除实用案例分析。

一、CRISPR/Cas9实现基因敲除的原理
CRISPR/Cas9系统是利用一段与靶序列互补的gRNA引导cas9核酸酶对特异靶向的DNA进行识别和切割,造成双链DNA断裂,然后细胞会利用自身具备的两种DNA修复机制对断裂的DNA进行修复,即非同源末端连接(Non-homologous end joining, NHEJ)或同源介导的修复(Homology-directed repair, HDR),而基因敲除则利用的是NHEJ的修复方式,在Cas9切割诱导DNA双链的断裂后,细胞启动NHEJ修复机制,在修复过程中通常会发生碱基插入或缺失的错配现象,如果插入或缺失3的非整数倍个的碱基,则会造成基因的移码突变,从而使靶基因无法编码正确的蛋白质,以此实现基因敲除效果,当然这是针对编码基因而言,对于非编码RNA的基因敲除或特定区域片段的删除,则需要在目的片段的两端分别设计一条gRNA,通过共转两条gRNA引导Cas9到指定位置切割达到删除目的片段的效果。
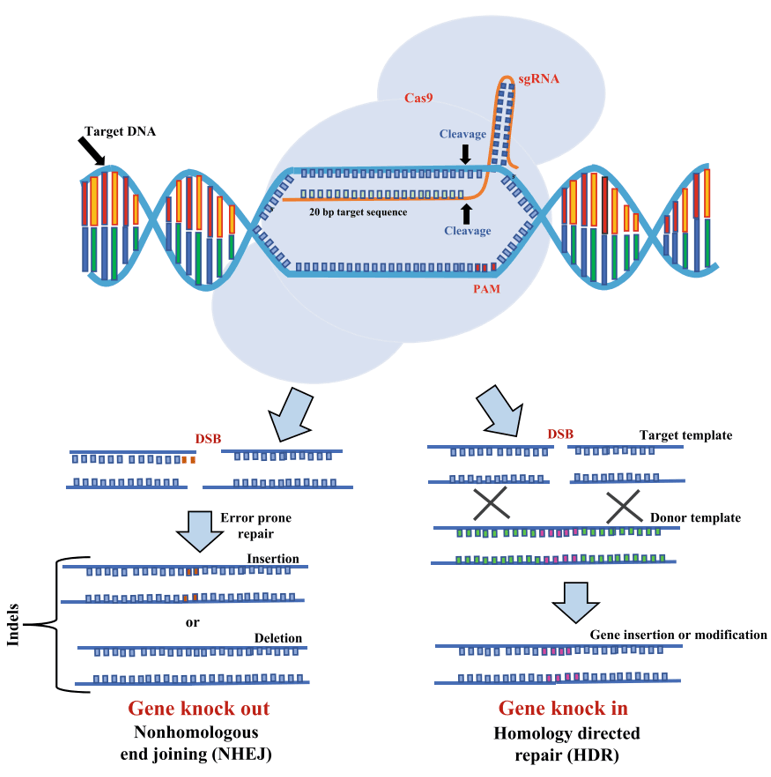
图1. CRISPR/Cas9介导的基因编辑示意图[1]
二、敲除单克隆细胞株的构建
1. 确定基因,选择合适的细胞系
根据研究内容确定待敲除的目的基因和细胞系。敲除前应首先通过网站查找预测、文献查阅等明确待敲除基因的功能并评估敲除此基因是否致死;在细胞系的选择上,优先选择容易转染且单克隆增殖能力较强的细胞。
2. gRNA的设计与合成
一般来说,基因特异的gRNA模板序列为位于PAM序列的前20nt。常用spCas9蛋白识别的PAM序列特征为NGG(其中N为任意核苷酸)。因此,gRNA模板序列选择非常方便,即使没有软件,我们也可根据序列自行设计。针对于gRNA序列的设计有很多网站可以参考,通过网站评分综合选择就好。这里小恒推荐两个较权威的网站:
① http://crispr-era.stanford.edu/index.jsp
② http://crispr.cos.uni-heidelberg.de/index.html
设计高效编辑的gRNA序列是敲除实验的关键,通常需要满足以下设计原则:
① gRNA序列应针对靶基因所有转录本的共有序列进行设计;
② 靶位点应设计在外显子上,在基因上游越靠前越好;
③ 尽量避开 GC 富集区域,以防止甲基化对互补配对的干扰;
④ gRNA应该以G 碱基开头,以提高转录效率;
⑤ 选择特异性位点,以降低脱靶概率。
需要注意的是,不同的gRNA靶点在基因敲除效率上有较大的差异,因此往往需要设计构建 2~3 个gRNA的基因敲除载体,再通过实验结果从中选出编辑效果最好的gRNA。
3. 合成gRNA
设计好与靶序列互补配对的gRNA序列后,我们需要将gRNA构建至相应的敲除载体上,目前常用的方法是利用限制性内切酶酶切形成粘性末端后连接gRNA,所以需要根据我们使用的Cas9-gRNA质粒的酶切位点选择合适的接头。举例说明,若我们选用BsmBI为酶切位点的载体,BsmBI的接头如下图2所示,
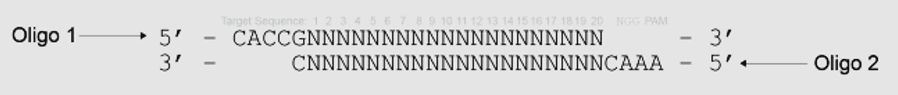
图2. BsmBI的接头设计
当我们锁定PAM序列(示例为针对spCas9的gRNA oligo设计)之后,只需取NGG前面20nt的序列,并设计如图所示的oligo,合成即可(新手小白们要注意合成方向哦)。

图3. BsmBI接头的oligo序列示例
4. 构建Cas9和gRNA表达载体
将合成的oligo进行退火,然后将其连接至经过酶切线性化的载体上(图4)。对于转染效率高的细胞,通常用质粒载体进行转染即可,对于较难转的细胞,通常选择构建到合适的病毒载体上包装成病毒再感染细胞以导入Cas9和gRNA。
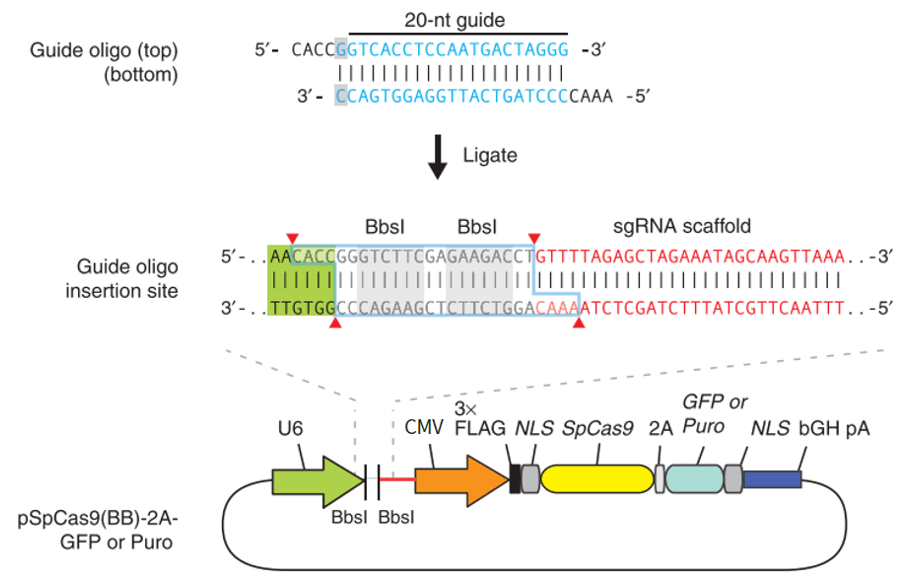
图4.构建Cas9和gRNA表达载体[2]
5. 筛选混合克隆,检测敲除效率
细胞转染表达载体48h后,可通过抗性或荧光标记筛选获得成功转入Cas9和gRNA的混合克隆细胞株,以提高获得单克隆的概率,随后抽提部分混合克隆的基因组通过PCR测序鉴定是否有套峰(图5),根据套峰的杂乱程度可以估算编辑效率,也可用T7E1错配酶酶切定量编辑效率(图6)。
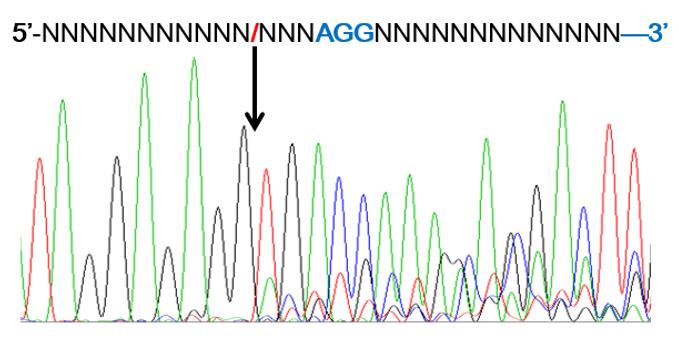
图5. PCR测序套峰示意图
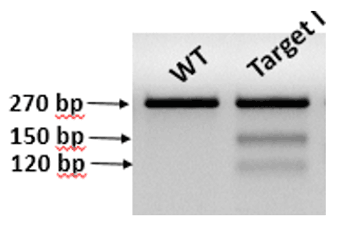
图6. T7E1酶切结果示意图
6. 挑取单克隆
将余下的混合克隆细胞有限稀释到96孔板中(293T、HeLa、MEF等细胞系推荐使用),稀释密度一般为50~90个细胞/板(图7),若编辑效率达50%以上,推荐分两块96孔板即可,最终选取20-30个单克隆以得到成功敲除的细胞系;也可以将细胞铺到100mm皿里,一周后挑取单克隆(注:这种方法适合单细胞不易成活的细胞系,但是挑取的单克隆偶尔不纯,有时需要重新亚克隆);若载体带有GFP等荧光标记,还可以通过流式细胞仪直接分选,把单个细胞种到孔板中直接获取单克隆。
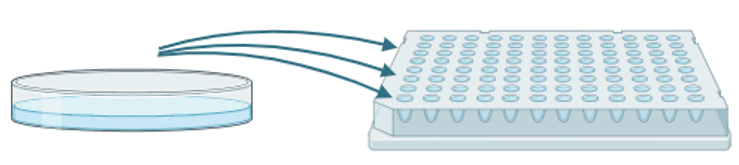
图7.单克隆的挑取
7. 单克隆的鉴定
单克隆长起来后就可以抽取基因组DNA或者提取总蛋白进行鉴定。常见的鉴定方法如下(图8):单克隆直接进行PCR测序,可以直观地看到编辑位点附近DNA序列的改变情况;WB鉴定,用抗体鉴定目的蛋白条带的有无,这是检测基因敲除效果最直接的方法,但是移码突变型的敲除可以表达出目的基因N端的部分肽段,如果所使用的抗体结合位点刚好在表达的残留蛋白上是可能被检测到的,另外,非特异性条带是蛋白检测中最常见的问题之一,使用高质量、特异性强的抗体是获得可靠结果的关键。
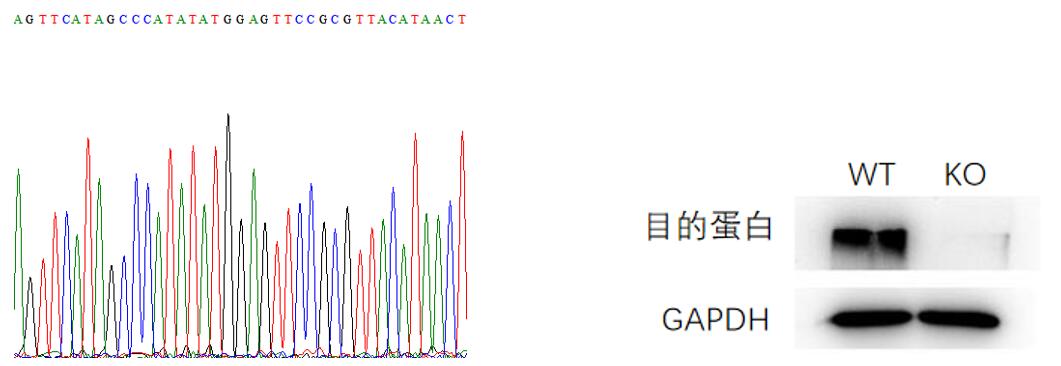
图8.单克隆的鉴定方法
三、片段敲除
对于非编码RNA敲除或特定基因组片段的删除,操作流程和以上相同,不同的是要在敲除片段的两端各设计一条gRNA,通过实验筛选两条有编辑效果的gRNA,两条gRNA和Cas9一起或分开同时转入细胞中,才能达到片段敲除的目的(图9),但是通常需要在片段两端前后100~300bp左右的位置来设计gRNA,所以这种方法只能达到非精准的片段敲除。如需精准敲除,建议使用敲入的方法。
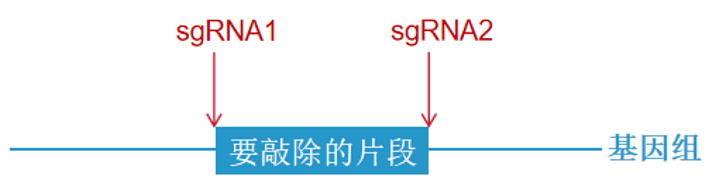
图9.片段敲除原理示意图
四、汉恒客户-敲除案例精选
【敲除案例1】编码基因的敲除
文章标题:Discovery of a potent allosteric activator of DGKQ that ameliorates obesity-induced insulin resistance via the sn-1, 2-DAG-PKCε signaling axis.
杂志:《Cell Metabolism》(IF=31.373)
敲除基因:DGKQ
载体工具: pHBcas9/gRNA-puro质粒
使用方法:使用Lipofectamine 2000在HepG2细胞中转染 pHBcas9/DGKQ/gRNA-puro 质粒,48 小时后,使用2 µg/ml嘌呤霉素筛选细胞3天。
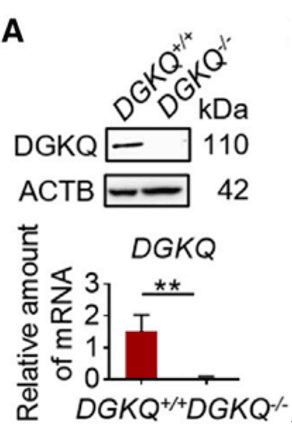
图10. HepG2中敲除 DGKQ(PMID:36525963)
【敲除案例2】特定区域DNA片段的删除
文章标题:Regulation of Hsa‐miR‐4639‐5p expression and its potential role in the pathogenesis of Parkinson's disease.
杂志:《Aging Cell》(IF=7.8)
敲除片段:hsa-miR-4639启动子片段
载体工具:pHB-gRNA-cas9-Zsgreen质粒
使用方法:将构建好的pHB-gRNA-cas9-Zsgreen质粒转染到SH-SY5Y细胞,转染后24h,用流式细胞术分选带绿色荧光的单克隆细胞到96孔板中,2-3周后进行单克隆鉴定。
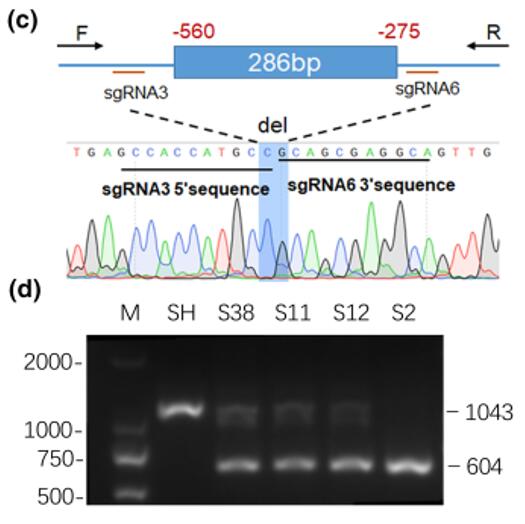
图11. SH-SY5Y细胞中敲除hsa-miR-4639启动子片段(PMID:37101349)
本期内容针对基因敲除原理、敲除单克隆细胞株的构建、基因片段敲除和敲除实用案例分析等四大方面进行了详细介绍,希望对基因敲除研究方向的小伙伴可以提供一些设计灵感。汉恒生物可以提供基因敲除相关的服务(包括敲除质粒构建、敲除病毒包装以及单克隆敲除细胞株筛选等服务),并拥有专业的技术服务团队为您答疑解惑,有意向的小伙伴欢迎与我们联系~下期我们将继续为大家介绍基因敲入,小伙伴们继续关注哦~~~!
参考文献
[1]Das T, Anand U, Pal T, Mandal S, Kumar M, Radha, Gopalakrishnan AV, et al. Exploring the potential of CRISPR/Cas genome editing for vegetable crop improvement: an overview of challenges and approaches. Biotechnol Bioeng. 2023;120:1215–1228. doi: 10.1002/bit.28344.
[2]Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 2013;8:2281–308. doi: 10.1038/nprot.2013.143.
[3]Zheng ZG, Xu YY, Liu WP, Zhang Y, Zhang C, Liu HL, Zhang XY, Liu RZ, Zhang YP, Shi MY, Yang H, Li P. Discovery of a potent allosteric activator of DGKQ that ameliorates obesity-induced insulin resistance via the sn-1, 2-DAG-PKCε signaling axis.Cell Metab.2023 Jan 3;35(1):101-117.e11.doi: 10.1016/j.cmet.2022.11.012.
[4]He L, Chen YM, Lin SZ, Shen RN, Pan H, Zhou YF, Wang Y, Chen SD, Ding JQ. Regulation of Hsa‐miR‐4639‐5p expression and its potential role in the pathogenesis of Parkinson's disease.Aging Cell.2023 Jun;22(6):e13840.doi: 10.1111/acel.13840.

查看更多

查看更多

查看更多

联系我们

返回顶部